Blog
Optunaからも使えるCMA Evolution Strategyの実装を公開しました。
Covariance Matrix Adaptation Evolution Strategy (CMA-ES)は、ブラックボックス最適化において最も有望な手法の1つです。CMA-ESはハイパーパラメータ最適化にも使われていて [1, 2]、近年では、ハイパーパラメータ最適化においても、評価回数が許容できる場合や並列化環境がある場合には、ベイズ最適化を上回る性能を示すことが報告されています[6]。
CMA-ESをPythonで実装しGitHubで公開したのでその使い方や性能について紹介します。Optunaからも利用できるようになっているのでぜひハイパーパラメータの最適化に使ってみてください。
CMA-ES
CMA-ESは多変量正規分布から解を生成し、その解の評価値を利用して、より良い解を生成するような分布に更新を行う手法です。変数間の依存関係のある問題や変数間のスケールが異なるような問題に対して有力であることが報告されています [3]。また、ある程度の評価回数を許容できる場合に、様々なブラックボックス最適化問題に対する実験において、100以上の最適化手法の中で最も良い性能を示したことが Loshchilovらによって報告されています[4]。
PythonライブラリとしてはCMA-ESの提案者Nikolaus Hansenが公開しているpycmaが最も有名です。Optunaにもpycmaとのintegration samplerが実装されています。しかしpycmaの設計は弊社の野村がNeurIPS 2019のMeta Learning workshopで発表した手法を実装する際にインターフェース上の問題があり、コード自体も複雑になってきてしまっている面があるため、今後のメンテナンス性を踏まえフロムスクラッチで実装しました。
CMA-ESのアルゴリズムについては、The CMA Evolution Strategy: A Tutorial [5]を参照してください。ユニットテスト等による実装の確認が難しいことから、matplotlib.animationベースの可視化ツールを実装しこちらで挙動を確認しながら実装を進めていきました。多変量正規分布の共分散行列が最適解付近で小さくなっていく様子が確認できます。

次のように変数間のスケールが異なる問題でも等高線に沿うような形で共分散行列が更新されていることが確認できます。

cmaesの使い方
それではライブラリの使い方を簡単に紹介します。PyPIにも上げているので、pipからインストールできます。
|
1 |
$ pip install cmaes |
このライブラリは2種類のインターフェースを提供しています。OptunaのSamplerとしてつかえるインターフェース (cmaes.sampler.CMASampler)と、pycmaのように ask() と tell() メソッドを提供する低レベルなインターフェース(cmaes.CMA) です。基本的には cmaes.sampler.CMASampler を用いてOptunaから利用することがおすすめです。Optuna samplerを使う場合は、次のようになります。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 |
import optuna from cmaes.sampler import CMASampler def objective(trial: optuna.Trial): x1 = trial.suggest_uniform("x1", -4, 4) x2 = trial.suggest_uniform("x2", -4, 4) return (x1 - 3) ** 2 + (10 * (x2 + 2)) ** 2 def main(): sampler = CMASampler() study = optuna.create_study(sampler=sampler) study.optimize(objective, n_trials=250) if __name__ == "__main__": main() |
注意点として、Categorical distributionは現状サポートしていません。Optunaが提供するpycmaベースのsamplerは、質的変数をサポートしていますが、label encodingにより連続空間に落としているため、カーディナリティの高い質的パラメーターが含まれる場合にはうまく最適化できないケースがあるかと思います。
そのためCMASamplerは、 trial.suggest_categorical を使っておらず、n_trialsを十分大きくできるときに利用してみてくださいい。trial.suggest_categoricalを使っている場合は基本的にデフォルトのTPE samplerを利用するのがおすすめです。
kurobakoとGitHub Actionsを使った継続的ベンチマーク
CMA-ESの実装の妥当性をユニットテスト等で検証するのが難しいことから、アルゴリズムの性能を確認できるベンチマークがあると非常に有用です。ベンチマークには ブラックボックス最適化ベンチマークツール「kurobako」の紹介 で解説されているkurobakoを利用しました。
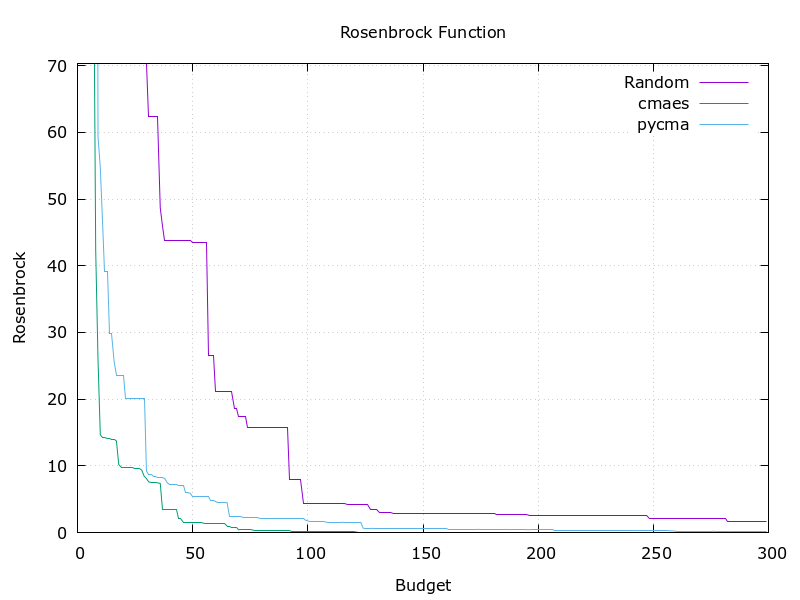
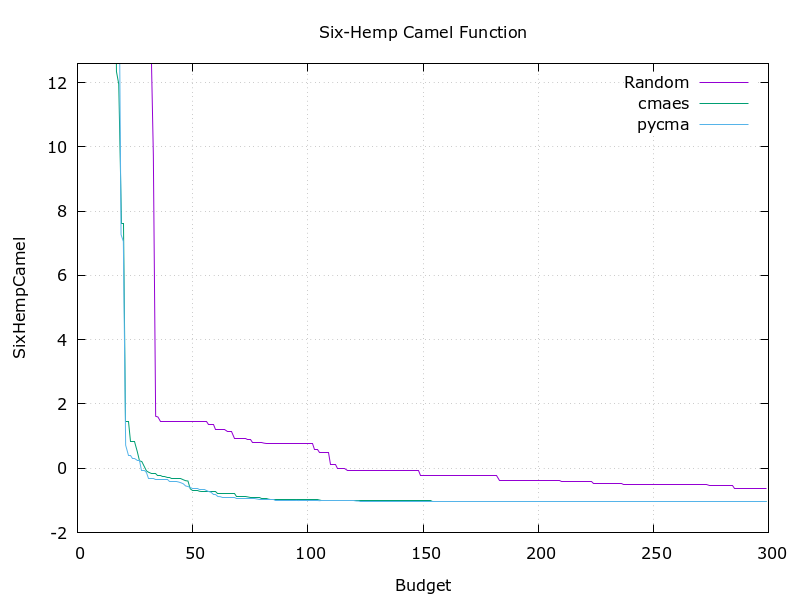
ベンチマーク関数はRosenbrock, Six-Hemp Camel, Himmelblauの3種類を用意しました。今回公開したCMA-ESの実装はアルゴリズム自体のコード行数が200行程度と比較的シンプルにおさまりましたが、その性能はpycmaと同程度であることが確認できています。
|
|
|
GitHub Actionsでの実行にはgithub-actioins-kurobakoというツールを作成・使用しました。Pull Requestを作成するとGitHub Actionsがkurobakoを実行し、その結果をPRにコメントすることで、ベンチマーク結果を継続的に追えるようにしています。諸事情でCyberAgent organizationはGitHub Actionsが使えないため自分の個人アカウントにforkしてこちらのPRのように動かして確認しています。
高速なOptuna sampler実装
CMA-ESの魅力的な点の1つは、次に探索すべきパラメーターの生成に必要な計算量が過去の試行回数に依存しない点です。GPやTPEでは代理モデルの構築時に過去の試行結果をすべて取り出してくる必要がありますが、CMA-ESで必要になるのは基本的に多変量正規分布の平均ベクトルと共分散行列の値だけです。
その一方でこちらのOptunaのIssueにあるようにOptunaが想定しているユースケースである分散最適化等を考慮すると、実装上悩ましい点がいくつかあります。現状、Optunaが提供しているpycmaのintegrationは試行回数が積み上がるにつれ必要な計算量が増加していく課題があります。
| trials | パラメーターの次元数 | pycma’s integration | this library |
| 100 | 10 | 6.000s | 0.706s |
| 500 | 50 | 4m 52.008s | 2.236s |
| 1000 | 100 | 37m 41.617s | 7.116s |
今回公開している実装では、多変量正規分布の平均ベクトルや共分散行列等をstorage上でキャッシュし、分布の世代番号(更新されるたびにインクリメントされる値)をチェックしながら最新分のtrialを追加適用しています。またOptunaの内部の関数も一部再実装しました。高速化についてはまたOptuna側にもPRを送っていけたらと思います。
おわりに
本記事ではOptunaから利用可能なCMA-ES実装の解説を行いました。シンプルな実装ながらpycmaと同程度の性能であることを確認し、Optunaから利用する際には比較的高速に動作するように実装しています。みなさんもぜひ使ってみてください。
参考文献
[1] Friedrichs, F. and Igel, C. Evolutionary Tuning of Multiple SVM Parameters.Neurocomputing, 64:107–117, 2005.
[2] Watanabe, S. and Le Roux, J. Black box optimization for au-tomatic speech recognition. In 2014 IEEE InternationalConference on Acoustics, Speech and Signal Processing (ICASSP), pp. 3256–3260, 2014.
[3] Rios, L. M. and Sahinidis, N. V. Derivative-free optimization: a review of algorithms and comparison of software implementations. Journal of Global Optimization, 56(3):1247–1293, 2013.
[4] Loshchilov, I., Schoenauer, M., and Sebag, M. Bi-population CMA-ES Algorithms with Surrogate Models and Line Searches. In Proceedings of the 15th annual conference companion on Genetic and evolutionary computation, pp. 1177–1184, 2013.
[5] N. Hansen. The CMA Evolution Strategy: A Tutorial. arXiv:1604.00772, 2016.
[6] Loshchilov, I. and Hutter, F. CMA-ES for Hyperparameter Optimization of Deep Neural Networks. In ICLR Workshop, 2016.
Author